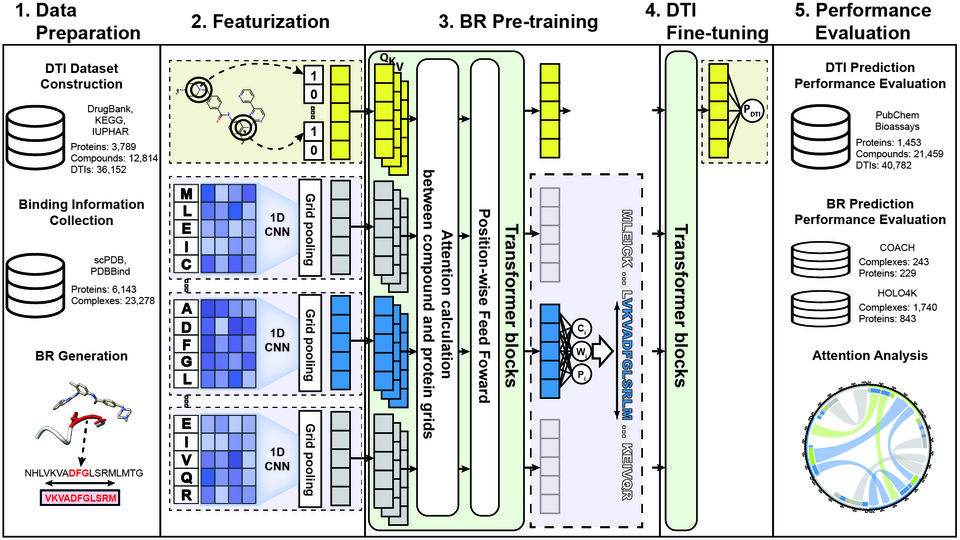
모델은 대규모의 단백질 3차원 구조 데이터베이스로부터 화합물과의 결합지역을 추출하여 컨볼루션신경망과 트랜스포머(Transformer) 기반의 딥러닝 모델로 단백질 서열상의 결합지역을 예측할 수 있도록 학습

전 세계적으로 수많은 제약회사들이 새로운 약물을 개발하기 위해 천문학적인 시간과 비용을 투자하고 있다.
여기에, 인공지능(AI) 기술을 활용하여 신약 후보물질 탐색 시간을 단축함으로써 결과적으로 신약개발에 소요되는 기간과 비용을 획기적으로 줄일 수 있어 혁명적 변화를 이끌 수 있다.
지스트(광주과학기술원, 총장 김기선) 전기전자컴퓨터공학부 남호정 교수 연구팀은 단백질 서열 기반으로 약물과 표적 단백질의 결합지역 및 상호작용을 예측(Highlights on Target Sequence 이하, HoTS) 하는 인공지능 기술을 개발했다.
그러나, 이번에 연구팀이 개발한 모델인 HoTS는 약물-표적 단백질이 결합하는 부분을 사전학습한 후 예측하게 함으로써, 높은 예측 정확도와 함께 약물-표적 단백질 상호작용 예측의 근거도 함께 제시하여 신약개발 연구자들에게 보다 신뢰할 수 있는 유효화합물 예측 결과를 제시해 줄 수 있게 되었다.
연구팀의 이 AI모델은 대규모의 단백질 3차원 구조 데이터베이스로부터 화합물과의 결합지역을 추출하여 컨볼루션신경망(CNN, Convolutional Neural Network)과 트랜스포머(Transformer) 기반의 딥러닝 모델로 단백질 서열상의 결합지역을 예측할 수 있도록 학습되었다.
결합지역을 학습한 후, 해당 학습을 기반으로 하여 더 많은 트랜스포머 계층을 통해 약물-표적 단백질 상호작용을 예측할 수 있으며, 그 결과 딥러닝 모델이 결합지역과 함께 약물-표적 상호작용을 예측할 수 있다.
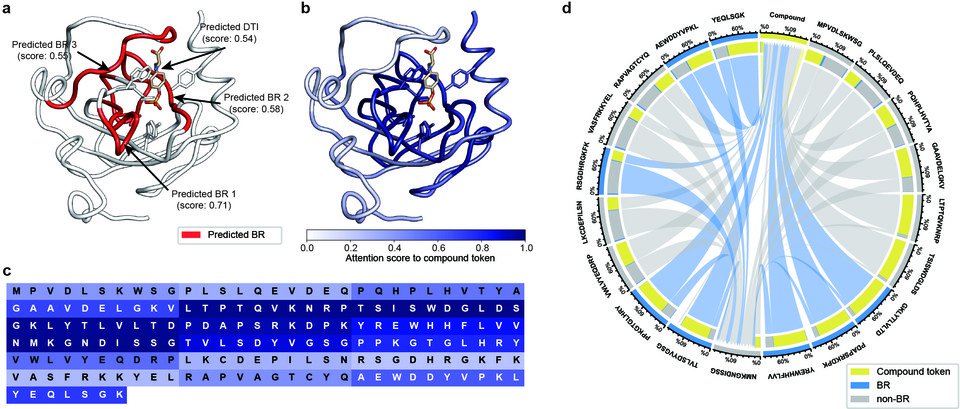
결과적으로 HoTS 모델은 다른 딥러닝 모델들보다 더 높은 예측력을 보여주었으며, 결합지역 예측도 단백질 서열 정보만을 사용함에도 불구하고 3차원 구조 기반의 타 예측 모델과 비슷한 수준의 성능을 확인하였다.
남호정 교수는 “본 연구성과는 신약 개발 단계 중 유효화합물 발굴의 효율성을 크게 높여주는 기술이며, 무엇보다 3차원 구조 정보가 없는 신규 표적 단백질에 대한 신약 개발의 가능성을 열어줬다는데 의의가 있다” 면서 “향후 해당 모델을 통해 약 개발 단계에서의 빠르고 효율적인 유효화합물 발굴이 가능할 수 있을 것으로 기대된다”고 말했다.
한편, 지스트 남호정 교수팀이 수행한 이번 연구는 ‘설명가능한 인공지능(XAI) 기반 약물 후보의 독성 및 부작용 예측 시스템 개발’, ‘지스트-전남대학교병원 공동연구과제’, 생명노화연구소’ 사업의 지원과 과학, 기술, 엔지니어링 및 의학 분야 글로벌 학술지 화학정보학 저널(Journal of Cheminformatics)에 '단백질 결합 영역 및 약물-표적 상호작용의 서열 기반 예측(Sequence-based prediction of protein binding regions and drug-target interactions-다운)'란 제목으로 지난 8일자 온라인 게재됐다.

