생명과학 분야의 신규 가설 발굴 도구로서의 인공지능 방법론과 구축된 모델은 암 등의 세포 상태에 특이적으로 나타나는 염색질 상호작용을 포착하는 방법론으로서 활용을 기대...

서울대학교 공과대학(학장 홍유석)은 컴퓨터공학부 김선 교수 연구팀이 DNA의 3차원적 접힘에 의한 상호작용을 포함하는 생물학적 요인들을 이용하여 유전자의 발현 조절을 모델링하는 트랜스포머(transformer) 기반 인공지능(AI) 모델인 크로모포머(Chromoformer)를 개발했다.
연구팀은 이 AI 모델을 통해 유전자 발현 예측의 성능이 기존 모델에 비해 향상되어 보다 효과적인 모델링이 가능함을 보였다. 뿐만 아니라 학습된 인공지능 모델 해석을 통해 간암 세포주의 특이적인 유전자 발현 관련 인핸서(enhancer) 활성화 포착이 가능하고, 전사 공장(transcription factory) 및 폴리콤 그룹 소체(Polycomb group body)와 관련된 조절 인자의 동역학을 포착할 수 있었다.
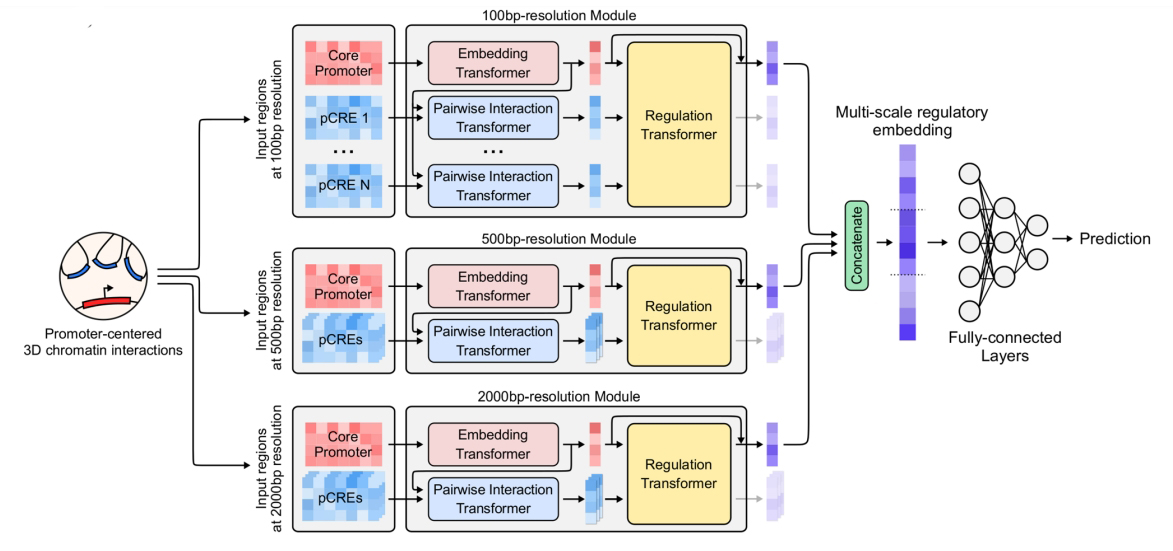
인공지능과 생명과학 분야 융합의 산물인 이번 연구는 인공지능 기술이 생명과학 분야의 새로운 가설 발굴을 위한 하나의 도구로서 활용될 수 있다는 새로운 연구 패러다임의 실질적인 예를 제시하였고, 구축된 모델은 암 등의 세포 상태에 특이적으로 나타나는 염색질 상호작용을 포착하는 방법론으로서 활용이 가능할 것으로 기대된다.
한편, 김선 교수와 BK21지능형컴퓨팅사업단 및 생명과학공동연구원 소속 이도훈 박사후연구원, 양지원 서울대 인공지능 융합과정이 참여한 이번 연구는 국제 학술지 네이처 커뮤니케이션스(Nature Communications)에 '트랜스포머를 사용하여 큰 게놈 창과 3차원 염색질 상호 작용으로 히스톤 코드 학습(Learning the histone codes with large genomic windows and three-dimensional chromatin interactions using transformer-아래 첨부)'란 제목으로 지난 5일 게재됐다.

