MIT의학공학과학연구소(IMES)‧인공지능연구소(CSAIL)-맥마스터대학 감염병 연구소 공동 연구팀의 이번 항생제 예측에 사용된 모든 사용자 지정 코드는 깃허브를 통해 오픈 소스로 공개했으며, 이 연구에서 사용된 복제된 스냅샷 역시 깃허브에서 확인할 수 있다.

지난 수십 년 동안 새로운 항생제는 거의 개발되지 않았으며, 새로 승인된 항생제는 기존 약물과 약간 다르다. 새로운 항생제를 선별하는 현재의 방법은 막대한 비용이 들고, 상당한 시간 투자가 필요하며, 일반적으로 좁은 범위의 화학적 다양성으로 제한된다.
국제보건기구(WHO)에 따르면 현재, 전세계적으로 항생제 내성으로 사망한 30여만명을 포함하여 약물 내성 질환으로 인해 매년 최소 70만 명 이상이 사망한다고 한다. 또 기존 항생제로 치료가 불가능한 변종 박테리아, 일명 '슈퍼박테리아'가 속속 등장하고 있는 상황이다.
여기에 인공지능(AI) 알고리즘을 사용하여, MIT와 캐나다 맥마스터대학교(McMaster University)의 공동 연구팀이 많은 약물 내성 감염을 일으키는 '슈퍼박테리아'류를 사멸시킬 수 있는 새로운 항생제를 발견했다.
이 물질은 병원에서 자주 발견되고 병원 문손잡이와 장비 등에서 장기간 생존할 수 있으며 폐렴, 뇌막염, 그리고 다른 심각한 감염으로 이어질 수 있는 슈퍼박테리아의 일종으로 가장 심각도가 높은 '아시네토박터 바우마니(Acinetobacter baumannii. 이하, 아시네토박터)' 퇴치에 도움이 될 수 있다. 이 아시네토박터는 이라크와 아프가니스탄 전장에서 부상당한 군인의 감염을 일으키는 주요 원인이기도 했다고 한다.
지난 수십 년 동안 많은 병원성 박테리아가 기존 항생제에 대한 내성이 증가한 반면 새로운 항생제는 거의 개발되지 않았다. 연구팀은 머신러닝 모델을 사용하여 약 7,000개의 잠재적 약물 화합물 라이브러리에서 이 물질을 식별한 것이다.
이 연구 이전인 불과 몇 년 전 MIT 의학공학과학연구소(IMES) 제임스 콜린스(James J. Collins) 생물학공학과 교수는 맥마스터 대학 감염병 연구소의 생화학 및 의과학과 조나단 M. 스톡스(Jonathan M. Stokes) 교수 및 MIT 컴퓨터과학 및 인공지능연구소(CSAIL) 레지나 바르질라이(Regina Barzilay) 교수는 등은 AI를 사용하여 증가하는 이 문제를 해결하기 시작했다.
이어 초기 시연(참고)에서 연구원들은 대장균의 성장을 억제할 수 있는 화학 구조를 식별하기 위해 머신러닝 알고리즘을 훈련했다. 1억 개 이상의 화합물에서 이 AI 알고리즘은 슈퍼 항생물질 '할리신(Halicin)' 을 발견했다. 그들은 이 물질이 대장균뿐만 아니라 치료에 내성이 있는 여러 다른 박테리아 종을 죽일 수 있음을 확인했다.
연구팀이 '할리신'이라고 부르기로 결정한 이 물질은 미국의 영화감독 스탠리 큐브릭(Stanley Kubrick)의 1968년 작 SF영화 '2001 스페이스 오디세이(2001 : A Space Odyssey)'의 가상 인공지능 시스템 '할(HAL)'의 이름을 따서 명명되었다.
스톡스 교수는 "우리는 그 논문 '항생제 발견에 대한 딥러닝 접근법(A Deep Learning Approach to Antibiotic Discovery-다운) 이후, 이러한 머신러닝 접근 방식이 복잡한 항생제 발견 작업에 효과적일 수 있다는 사실을 확인한 후, 다양한 약제에 내성을 가진 세균이나 바이러스를 말하는 다제내성(Multiple Drug Resistance, MDR) 박테리아 감염의 공공의 적 1호로 인식되는 아시네토박터에 주목했습니다"라고 말했다.
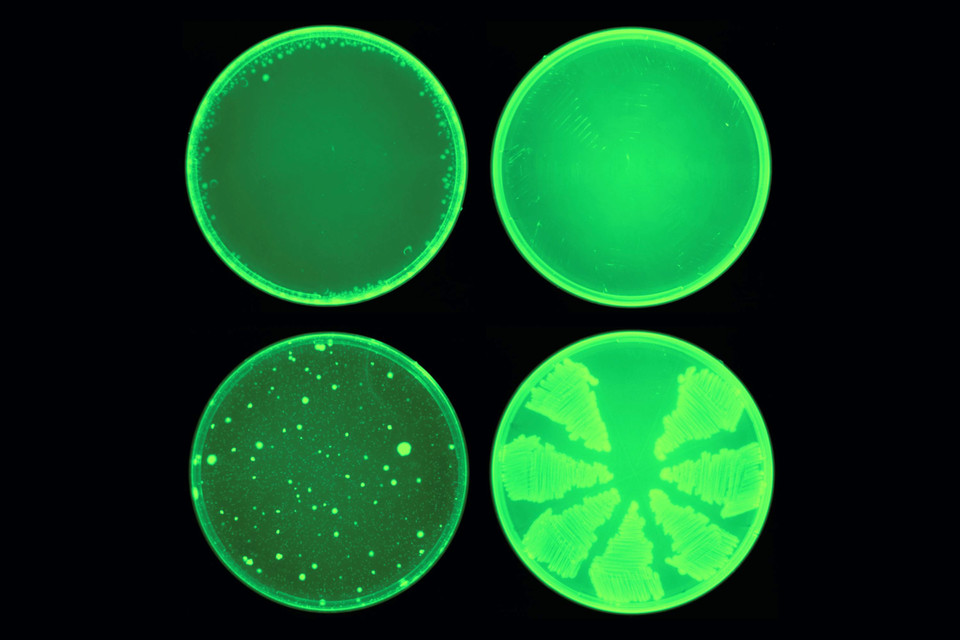
다시 본론으로 돌아와서, MIT와 맥마스터대학 공동연구팀은 계산 모델에 대한 훈련 데이터를 얻기 위해 먼저 실험실 배양으로 자란 아시네토박터를 약 7,500개의 서로 다른 화합물에 노출시켜 어떤 화합물이 미생물의 성장을 억제할 수 있는지 확인했다. 그런 다음, 각 분자의 구조를 모델에 입력했다. 또한 각 구조가 박테리아 성장을 억제할 수 있는지 여부를 모델에 인지시켰다. 이를 통해 알고리즘은 성장 억제와 관련된 화학적 특징을 학습할 수 있었다.
이후 연구팀은 모델을 사용하여 MIT와 하버드대학의 브로드연구소(Broad Institute)의 약물용도 변경 허브(Drug Repurposing Hub-보기)에서 이전에 본 적이 없는 6,680개의 화합물 세트를 분석했는데, 2시간도 채 걸리지 않은 이 분석에서 상위 화합물이 도출되었다. 연구진은 이 중 기존 항생제나 학습 데이터의 분자와 다른 구조를 가진 화합물을 중심으로 240개를 선별하여 실험실에서 테스트했다.
연구팀은 이 실험을 통해 매우 강력한 항생제 한 가지(아바우신)를 포함하여 9가지 항생제를 도출시켰다. 이 물질은 원래 잠재적인 당뇨병 치료제로 연구되었던 이 화합물로 특히, 아시네토박터를 사멸시키는 데 매우 효과적이면서 녹농균, 황색포도상구균, 카바페넴 내성 장내세균(Carbapenem-resistant Enterobacteriaceae, CRE) 등 다른 박테리아 종에는 아무런 영향을 미치지 않는 것으로 확인했다.
이처럼 좁은 스펙트럼에서의 살상 능력은 박테리아가 약물에 대한 내성을 빠르게 확산시킬 위험을 최소화하기 때문에 항생제에서 바람직한 특징으로 또 다른 장점은 이 약물이 사람의 장에 서식하는 유익한 박테리아를 살리고 클로스트리듐 디피실리균(Clostridium difficile)과 같은 건강한 사람에게는 감염 증상을 유발하지 않지만 극도로 쇠약하거나 면역기능이 감소된 사람에게 증상을 일으키는 기회 감염(Opportunistic Infection)을 억제하는 데 도움이 될 수 있다는 점이다.
특히, 연구팀은 생쥐를 대상으로 한 연구에서 '아바우신(abaucin)'이라고 명명한 이 약물이 아시네토박터 감염을 치료할 수 있음을 보여주었으며, 실험실 테스트에서 인간 환자로부터 분리한 다양한 약물 내성 아시네토박터 균주에 대해서도 효과가 있음을 확인했다고 밝혔다.
또한 추가 실험에서 이 약물은 세포가 세포 내부에서 세포 외피로 단백질을 운반하는 데 사용하는 ‘지단백질 밀매(lipoprotein trafficking)’로 알려진 과정을 방해함으로써 세포를 죽인다는 사실을 밝혔다. 구체적으로, 이 약물은 이 과정에 관여하는 단백질인 LolE를 억제하는 것이다.
모든 그람 음성 박테리아는 이 효소를 발현하기 때문에 연구팀의 새로운 항생물질 아바우신이 아시네토박터를 표적으로 삼는 데 매우 선택적이라는 사실에 놀랐다고 한다. 스톡스 교수는 "우리는 아직 실험 데이터 수집을 완료하지는 못했지만, 아시네토박터가 다른 그람 음성균과는 조금 다르게 지단백질(lipoprotein)을 운반하기 때문이라고 생각합니다"라며, "그래서 이렇게 좁은 스펙트럼의 활동을 보이는 것으로 보입니다"라고 말했다.
연구팀은 현재, 맥마스터대학의 다른 연구자들과 협력하여 이 화합물의 의학적 특성을 최적화하여 궁극적으로 환자에게 사용할 수 있도록 개발하기 위한 연구에 경진하고 있다. 또한, 이 모델링 접근법을 사용하여 황색포도상구균과 녹농균으로 인한 감염을 포함한 다른 유형의 약물 내성 감염에 대한 잠재적 항생제를 식별할 계획이다.
한편, 연구팀의 이번 연구는 국제 학술지 네이처 케미컬 바이올로지(Nature Chemical Biology)에 "딥러닝을 이용한 아시네토박터 바우마니균 표적 항생제 발견(Deep learning-guided discovery of an antibiotic targeting Acinetobacter baumannii-보기)'란 제목으로 지난 25일(현지시간) 게재됐다.
특히, 연구팀의 이번 항생제 예측에 사용된 모든 사용자 지정 코드는 깃허브(다운)를 통해 오픈 소스로 공개했으며, 이 연구 논문에서 사용된 복제된 스냅샷 역시 깃허브(다운)에서 확인할 수 있다.

