새롭게 개발한 딥러닝 모델은 높은 정확성을 보인다. 의료 영상 분석에서 분할 결과의 정확성을 평가하는 DSC 점수는 75~78%를 달성, 인접한 세포핵을 잘 구분했는지를 비교하는 AJI 성능의 경우 55~62%의 우수한 성능을

암 환자를 진단하고 그 예후를 예측하기 위해서는 병리영상 속 세포핵들의 모양을 확인하고 개수를 세는 작업이 필수다. 그러나 높은 성능의 딥러닝 모델을 학습하기 위해서는 정확하게 그려진 세포핵 데이터가 필요하다.
세포핵의 형태는 다양하며, 한 영상에서는 수십만 개 이상의 세포핵들이 존재하기 때문에 이를 일일이 데이터화 하는 것은 시간과 비용이 많이 소요된다.
이 문제를 해결하기 위해 최근에는 세포핵의 중심에 점을 찍어 학습데이터로 사용하는 ‘약지도 학습 기반’의 딥러닝 모델 연구가 활발히 이루어지고 있다. 이는 기존의 데이터셋 구축 방식을 간결하게 만들었지만 세포핵의 경계면에 대한 정보를 알 수 없어 붙어있거나 겹쳐있는 세포핵을 정확히 영역화하지 못할뿐더러, 세포핵의 정중앙에 위치해야만 학습이 잘 된다는 한계가 있었다.
이에 DGIST(총장 국양) 로봇및기계전자공학과 박상현 교수팀이 병리학 영상에서 세포핵을 정확하게 분석하는 새로운 인공지능 기술을 개발했다. 대략적인 세포핵 레이블링 만으로 겹쳐있는 세포핵을 정확하게 구분할 수 있어, 병리영상 미세환경 분석에 큰 기여를 할 수 있을 것으로 기대된다.

박상현 연구팀은 딥러닝 모델에 방향장 모듈과 중심점 예측 모듈을 추가해 세포핵을 더 정확하게 구분하도록 개선했다. 또한, 점이 세포핵 내부에만 존재한다면 영역화가 가능하도록 Expectation-Maximization 알고리즘을 도입하여 불확실한 점 레이블을 중앙으로 조정할 수 있는 과정을 추가했다.
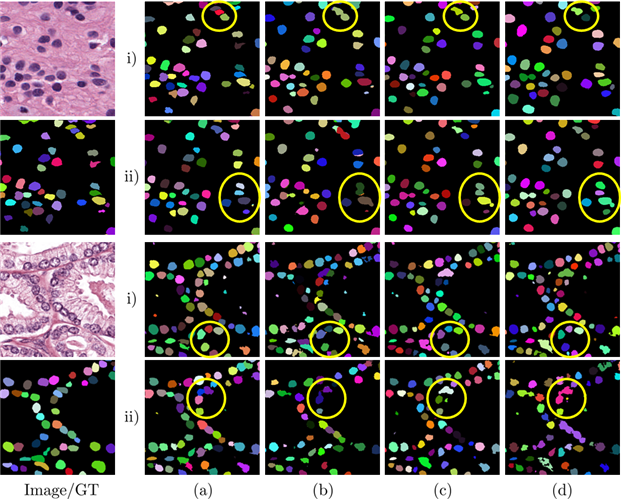
이 기술을 통해 세포핵을 정확하게 영역화하고 세포핵 간의 경계면을 찾을 수 있게 되었다.
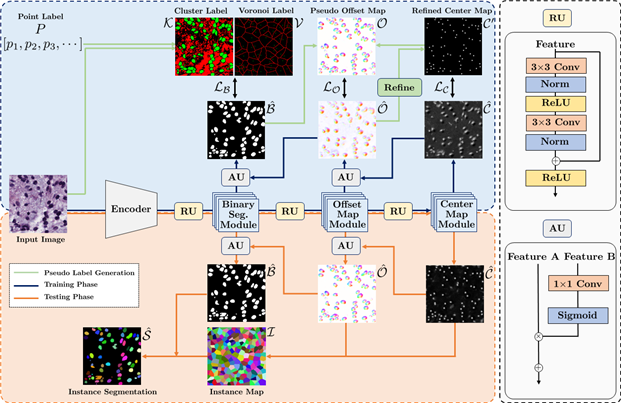
연구팀이 새롭게 개발한 딥러닝 모델은 높은 정확성을 보인다. 의료 영상 분석에서 분할 결과의 정확성을 평가하는 DSC(Dice similarity coefficient) 점수는 75~78%를 달성했으며, 인접한 세포핵을 잘 구분했는지를 비교하는 AJI(Aggregated Jaccard Index) 성능의 경우 55~62%의 우수한 성능을 보였다.
특히, 점이 정중앙에 존재하지 않는 경우 AJI 성능은 기존기법들에 비해 11~14% 향상된 성능을 보였다.
로봇및기계전자공학과 박상현 교수는 "이번 연구를 통해 데이터셋을 구축하는데 소요되는 시간과 비용을 크게 절감하면서도 세포핵을 정확하게 분석하는 것이 가능해졌다"며, "이 기술이 병리영상을 분석하여 환자의 질병을 진단하고 예후를 예측하는 데 큰 기여를 할 것으로 기대된다"고 말했다.
한편, 이번 연구는 결과는 지난 8일부터 10일까지 캐나다 벤쿠버에서 개최된 인공지능 분야 최상위 학회인 MICCAI(Medical Image Computing and Computer Assisted Intervention)에서 '프로넷: 핵 인스턴스 세분화를 위해 형상 유도 오프셋 맵을 사용한 포인트 세분화(PROnet: Point Refinement using Shape-guided Offset Map for Nuclei Instance Segmentation-아래 첨부)'란 제목으로 발표됐다.

